(30science.com) – Roma, 6 ott. – Si chiama Cell access through RNA sensing by Endogenous ADAR, o CellREADR, è un nuovo strumento di editing basato sull’RNA che prenda di mira le singole cellule piuttosto che i geni. Descritto sulla rivista Nature, questo risultato è stato raggiunto dagli scienziati della Duke University. Questo approccio, spiegano gli autori, permetterebbe di prendere di mira con precisione qualunque tipo di cellula, andando a fornire determinati enzimi o proteine nel luogo bersaglio. Il team, guidato da Z. Josh Huang e Yongjun Qian, ha utilizzato una sonda a base di RNA, dimostrando la possibilità di introdurre nelle cellule delle etichette fluorescenti per marcare tipi specifici di tessuto cerebrale. Il sistema di monitoraggio e controllo cellulare selettivo si basa sull’enzima ADAR, presente in nelle cellule animali.
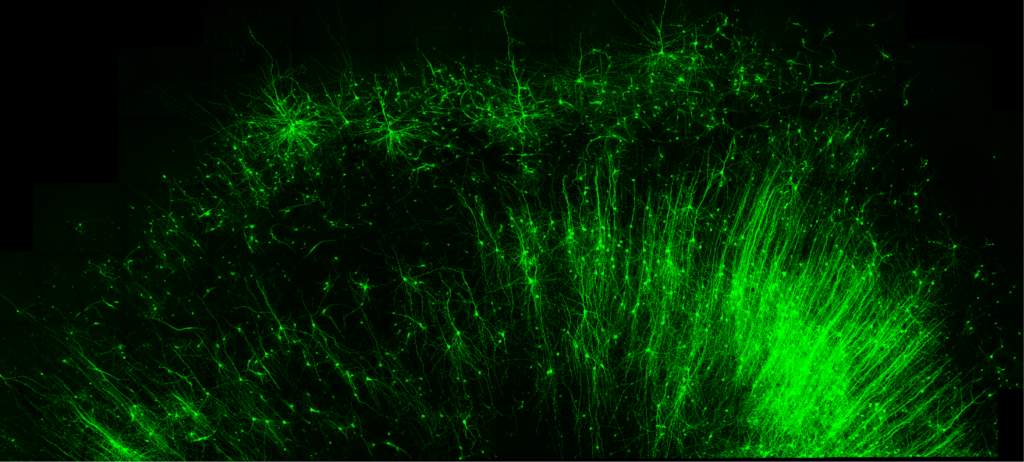
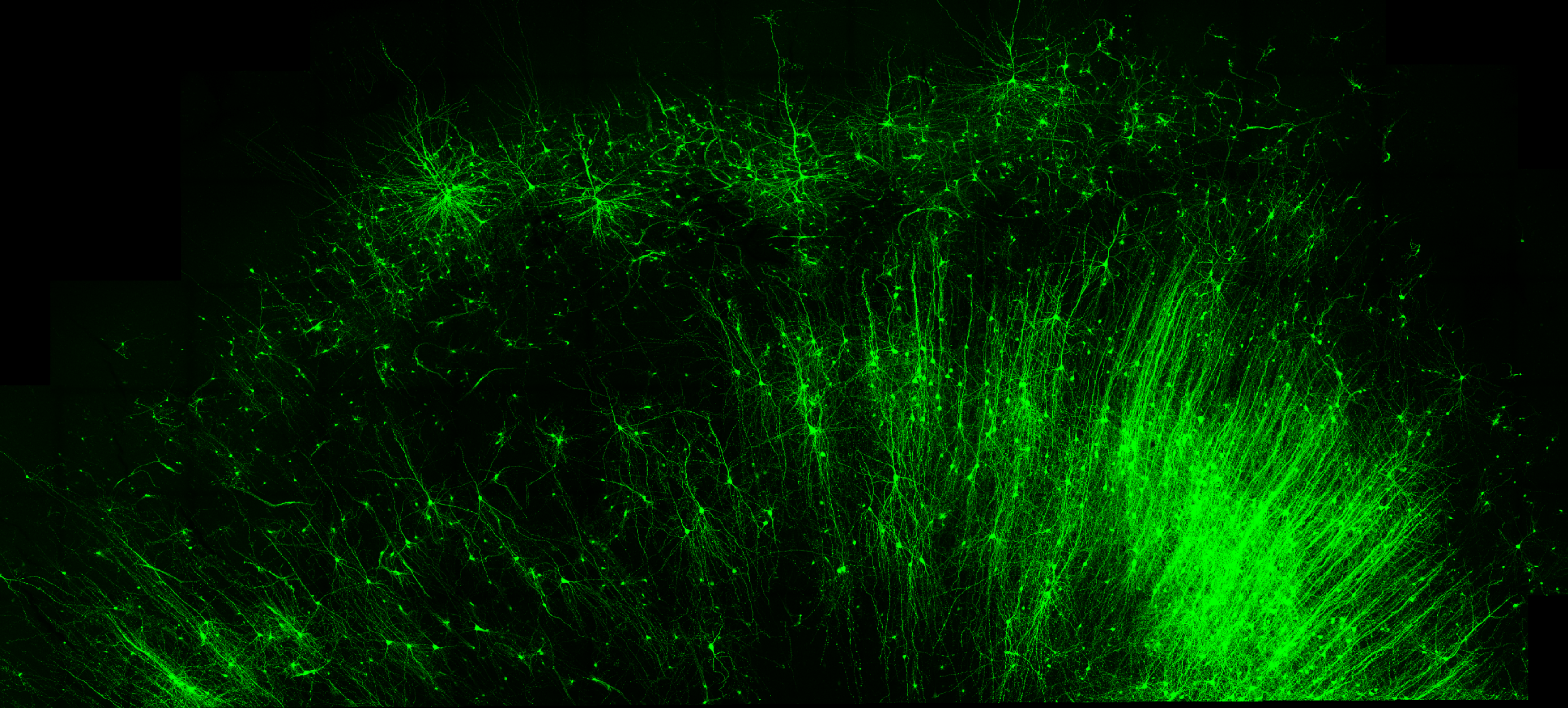
Contrassegnare e illuminare solo le cellule “freno” inibitorie (verdi) nel tessuto cerebrale umano è solo una delle tante cose che il nuovo strumento della Duke University, CellREADR, può fare. CREDITO Derek Southwell, Duke University
“La tecnologia – CellREADR afferma Huang – potrebbe avere moltissime applicazioni e potrebbe funzionare in tutto il regno animale. Questo fornisce una tecnologia semplificata, scalabile e generalizzabile per monitorare e manipolare tutti i tipi di cellule animali. È uno strumento davvero rivoluzionario nella gestione della malattia”. CellREADR, spiegano gli esperti, è una stringa personalizzabile di RNA composta da tre sezioni principali: un sensore, un segnale di stop e una serie di progetti. Una volta identificato l’RNA bersaglio della cellula che si vuole studiare, lo strumento viene calibrato sul filamento complementare del materiale genetico. Questo si unisce alla cellula selezionata e attiva l’enzima ADAR per modificare i nucleotidi nel codice genetico. L’enzima ADAR è un meccanismo di difesa cellulare progettato per modificare l’RNA a doppio filamento. Il segnale di stop impedisce la creazione di progetti proteici e viene rimosso dopo che il sensore si aggancia alla sequenza bersaglio. “Siamo stati in grado di dimostrare il funzionamento di questo sistema – commenta Huang – CellREADR è stato utilizzato per etichettare specifiche popolazioni di cellule cerebrali nei topi viventi e di tessuti umani raccolti da pazienti epilettici”. “Grazie a CellREADR – conclude Derek Southwell, altra firma dell’articolo – possiamo scegliere le popolazioni da studiare e iniziare davvero a studiare l’intera gamma di tipi cellulari presenti nel cervello umano. Questo strumento ci aiuterà a sviluppare nuove terapie per i disturbi neurologici e una serie di altre condizioni”. (30science.com)